
În secvențe genomice bacteriene Cheia este găsită pentru a ști dacă acestea pot fi combatete cu anumite antibiotice. Având în vedere importanța lor pentru sănătatea publică, profilurile lor au fost grupate în prima bibliotecă spaniolă de bacterii rezistente la antibiotice.
Baza de date digitală, cu aproximativ 500 de bacteriia fost publicat de Centrul Național de Analiză Genomică (CNAG), Roche Diagnostics și Complexul Spital Universitar A Coruña-Institutul de Cercetare Biomedicală A Coruña (INIBIC), grație unei noi metodologii care permite obținerea mai rapidă și pe o durată a genomului bacterian complet. scară largă.
Noua bază de date digitală completează profilul genomic al acestor bacterii cu date clinice, geografice și microbiologice și cu alte studii online legate de rezistența antimicrobiană.
Acest lucru face posibilă monitorizarea de rutină a modelelor de transmisie ale acestei rezistențe. Conform Tyler Aliotoautor al studiu și liderul echipei CNAG, „caracterizarea completă a bacteriilor poate fi realizată numai cu ansambluri complete de genom care oferă cromozomul principal, în plus față de setul de plasmide și numărul de copii ale acestora”.
„Pentru a demonstra că acest lucru se poate face la scară largă, am secvențiat 500 de bacterii rezistente la antibiotice cu o combinație de tehnologii genomice de ultimă generație și am dezvoltat un flux de lucru automat pentru procesarea datelor”, adaugă el.
Portalul inCREDBle, care este numele această bibliotecă de ADN Disponibil pentru consultare, explorare și descărcare, compila un total de 461 de tulpini bacteriene rezistente la antibiotice, colectate de la 41 de spitale situate în 13 regiuni diferite ale Spaniei.
Noua bază de date digitală completează profilul genomic al acestor bacterii cu date clinice, geografice și microbiologice, precum și cu alte studii online legate de rezistența la antimicrobiene, devenind cea mai completă resursă pentru studiul Enterobacteriaceae, una dintre cele mai rezistente familii la antibiotice.
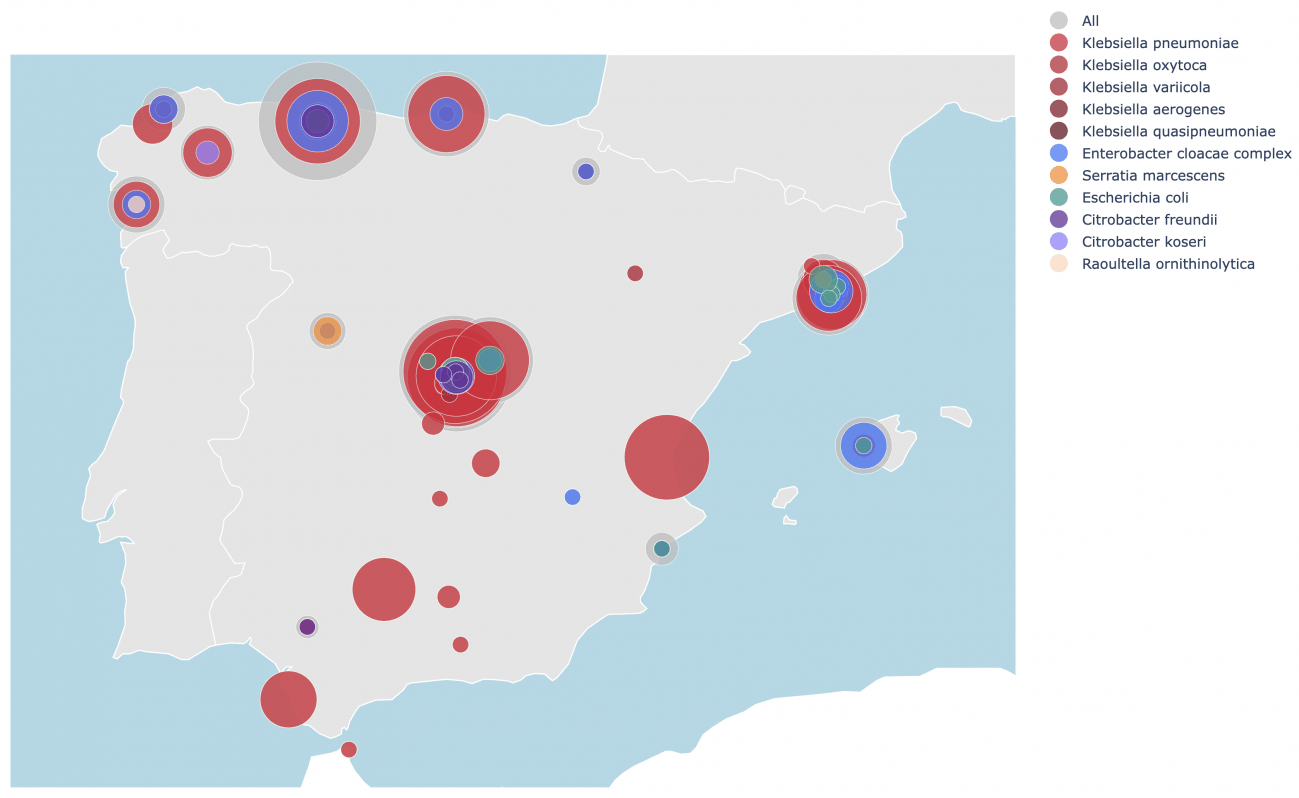
Distribuția bacteriilor rezistente la antibiotice după localizarea geografică la nivel național. /CNAG
Cheia tratamentelor viitoare
Obiectivul principal este de a oferi mai multe informații cu privire la mecanismele prin care speciile din ordinul Enterobacterales dobândesc sau dezvoltă această rezistență la medicamente, precum și mecanismele de bază de diseminare.
„Una dintre strategiile propuse de Centrul pentru Controlul și Prevenirea Bolilor (CDC) din Statele Unite pentru supravegherea rezistenței antimicrobiene este monitorizarea agenților patogeni rezistenți sau multirezistenți la medicamente. Tocmai baza de date genomică pe care am creat-o permite identificarea, localizarea și monitorizarea bacteriilor gram-negative care poartă unul dintre cele mai periculoase mecanisme de rezistență la antibiotice, enzimele numite carbapenemaze”, spune el. German Bouautorul studiului.
„Lucrarea poate fi folosită pentru studii viitoare cu un număr mai mare de tulpini și care includ o diversitate mai mare de agenți patogeni rezistenți. Acest lucru ar permite informarea dinamică, în timp real, cu privire la transmiterea microorganismelor multirezistente în țara noastră”, subliniază Bou, șeful INIBIC.

Lucrarea poate fi folosită pentru studii viitoare cu un număr mai mare de tulpini și care includ o diversitate mai mare de agenți patogeni rezistenți. Acest lucru ar permite informarea dinamică, în timp real, cu privire la transmiterea microorganismelor multirezistente în țara noastră.
Germán Bou, autor

Studiul oferă date noi legate de structura plasmidelor, tocmai datorită rolului lor important în transmiterea rezistenței antimicrobiene. O înțelegere profundă a acestor mecanisme ar putea ajuta la producerea unor tratamente mai eficiente care să evite o astfel de rezistență.
„Baze de date ca aceasta sunt foarte importante pentru a înțelege genetica și biologia cu care unele dintre aceste bacterii acționează împotriva antibioticelor. Sunt un instrument foarte valoros pentru cercetare; „Ei ajută la dezvoltarea de noi metode de diagnosticare și terapii cu impact mare pentru aceste microorganisme periculoase”, continuă el. Miguel Álvarez-Tejadode la Roche Diagnostics.
Principalele amenințări la adresa sănătății publice
Bacteriile rezistente la antibiotice, care provoacă infecții respiratorii și urinare, au devenit o problemă de sănătate publică la nivel mondial datorită capacității lor de a dezvolta rezistență la cele mai eficiente antibiotice de până acum, carbapenemele.
Potrivit Organizației Mondiale a Sănătății (OMS), ca urmare a rezistenței la medicamente antibioticele devin ineficiente și infecțiile devin dificil sau imposibil de tratat.
Potrivit OMS, ca urmare a rezistenței la medicamente antibioticele devin ineficiente și infecțiile devin dificil sau imposibil de tratat.
Apariția și răspândirea agenților patogeni rezistenți la medicamente amenință capacitatea noastră de a trata infecțiile obișnuite și de a efectua proceduri care salvează vieți, cum ar fi chimioterapia cancerului, operațiile cezariane, protezele de șold, transplanturile de organe și alte intervenții chirurgicale.
Prezentul studiu, care oferă prima bază de date de bacterii rezistente la antibiotice din Spania și o nouă metodologie genomică pentru a genera profilul ADN al acestor bacterii, contribuie la planul de acțiune promovat de OMS pentru a face față acestei amenințări globale, legată de consolidarea baza de cunoștințe și dovezi prin supravegherea și cercetarea acestor bacterii.
Referinţă:
Alioto, Tyler S., et al. „Dezvoltarea unui nou flux de lucru simplificat (AACRE) și a unei baze de date (inCREDBle) pentru analiza genomică a enterobacteriilor rezistente la carbapenem”. Genomica microbiană2023.
Fântână: SINCRONIZARE
Drepturi: Creative Commons.